注:この記事は、有識者個人の意見です。COVID-19有識者会議の見解ではないことに留意ください。
- 新型コロナウイルス(SARS-CoV-2)は、野生コウモリ由来のウイルスが人獣共通感染によって直接あるいは間接的にヒトに飛び火したものと考えられる。真の祖先ウイルスが何であり、どうやってヒトに飛び火したのかを解明するにはさらなる研究が必要である。
- SARS-CoV-2の人工ウイルス説や陰謀説にはそれなりの背景があるものの、客観的証拠は無い。
- ウイルスが変異するのは自然の摂理であり、SARS-CoV-2の変異株が伝播性の上昇や免疫効果の減弱を示すのも必然的な現象である。断片的な情報で一喜一憂するのではなく、変異株の病原性に関する適切な情報共有や客観的評価が重要である。
- SARS-CoV-2の変異は必ずしも強毒化に繋がるものではなく、肺炎ウイルスから風邪ウイルスへの進化に繋がっている可能性も少なくない。既知の風邪コロナウイルスに関する国内外の研究に照らすと、SARS-CoV-2が風邪コロナウイルスとして人類社会に定着していく蓋然性は高いように思われる。
- SARS-CoV-2やその変異株を肺炎ウイルスとしていつまでも特別視するのか、風邪コロナウイルスと認定するのか、純粋な科学的根拠に基づいて客観的に判断することでコロナ禍の出口戦略も見えてくる。
はじめに
2019年12月1日 - 新型コロナ感染症の最初の患者が中国の武漢で原因不明の肺炎を発症した日とされている[1]。その後、同様の患者が武漢で増え続け[1]、12月31日には世界保健機関(WHO)に正式に報告された[2]。その間、中国の研究グループは、12月30日に採取した患者検体の解析により、原因は新型コロナウイルスであることを明らかにし、その全ゲノム配列を解読して、2020年1月10日頃までに国際的データベース(GISAID)に登録している[1, 3]。この新型コロナウイルスのゲノム配列は、中国の広州に端を発して2002~3年に流行した重症急性呼吸器症候群(severe acute respiratory syndrome; SARS)の原因となったSARSコロナウイルス[4]やそれに近縁のコウモリコロナウイルス[5]に類似していることも中国から報告された[3]。これにより、国際ウイルス分類委員会(International Committee on Taxonomy of Viruses; ICTV)は新型コロナウイルスをSARSコロナウイルス2型(SARS-CoV-2)と命名した。WHOは、SARS-CoV-2感染症の正式名称をcoronavirus disease 19(COVID-19)と定め、3月11日にパンデミックを宣言した[2]。本稿の執筆時点(2021年12月)で、世界の累計感染者は約2億7千万人で、死者は520万人を超えている。日本でも173万人の感染者と18,000人を超える死者が出ており、社会生活や経済に甚大な影響が及んでいる。
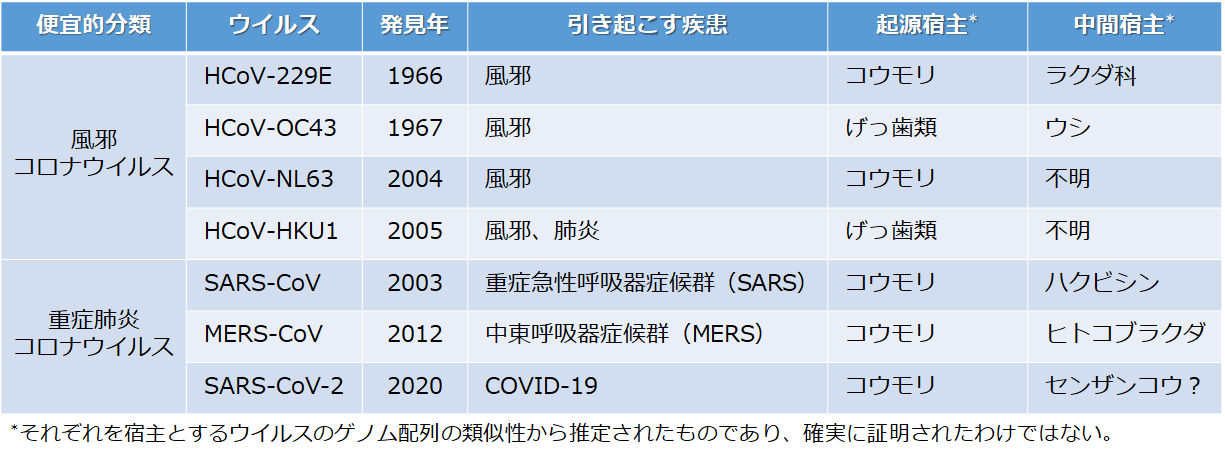
本稿では、SARS-CoV-2の由来や変異について基礎ウイルス学の視点から概説すると共に、今後このウイルスがどうなっていくのかについても推測してみたい。SARS-CoV-2以外のヒトコロナウイルスについても言及するので、それらについて【表1】にまとめておく。必要に応じてご参照いただきたい。
| 表1 |
| ヒトの病原コロナウイルスとその由来 |
 |
| Proc Soc Exp Biol Med. 1966 Jan;121(1):190-3.Proc Natl Acad Sci U S A. 1967 Dec;58(6):2268-73.N Engl J Med. 2003 May 15;348(20):1967-76.Nat Med. 2004 Apr;10(4):368-73.J Virol. 2005 Jan;79(2):884-95.N Engl J Med. 2012 Nov 8;367(19):1814-20.N Engl J Med. 2020 Feb 20;382(8):727-733.Int J Biol Sci. 2020 Mar 15;16(10):1686-1697. |
新型コロナウイルスはどこから来たのか?
動物のウイルスが人間に飛び火
出身地は「コウモリ」?
2002年、中国の広州で重症肺炎SARS(致死率10%)が発生し、香港を経由して世界各地に広がって行った。原因として同定されたのはSARSコロナウイルス(SARS-CoV)[4]であり、ゲノム配列のよく似たウイルスが広東省深圳市の食品市場で売られているハクビシンなどの小動物から見つかった[6]。その後、野生のコウモリからも類似のウイルス(SARS-like coronavirus; SL-CoV)がいくつか見つかっている[5, 7]。従って、SARS-CoVは、コウモリに感染していたコロナウイルスが自然界でハクビシンなどに伝播し、それが食品市場などでヒトに感染した人獣共通感染ウイルスと考えられる【表1】。同様に、中東呼吸器症候群(middle east respiratory syndrome; MERS、致死率35%)の原因として2012年に発見されたMERSコロナウイルス(MERS-CoV)[8]も、コウモリからヒトコブラクダを経て人に伝播した人獣共通感染ウイルスと考えられている【表1】。また、風邪の原因となることが知られているコロナウイルスは今までに4種類見つかっているが、それらについても、類似ウイルスが他の哺乳動物から見つかっており、人獣共通感染によってヒトに伝播した可能性が高い【表1】。
SARS-CoV-2は、ヒトの病気の原因になることが証明された7番目のコロナウイルスである。そのゲノム配列はSARS-CoVと79.6%の類似性を示すが、コウモリ由来のSL-CoVとは89.1%という高い類似性を有することがわかった[3]。さらに、雲南省の野生コウモリから分離されたRaTG13というコロナウイルスとは96.1%というさらに高い類似性が示された[9]。従って、SARS-CoV-2の出現には、何らかの形でコウモリが関わっていると推定される。
経由地は「センザンコウ」?
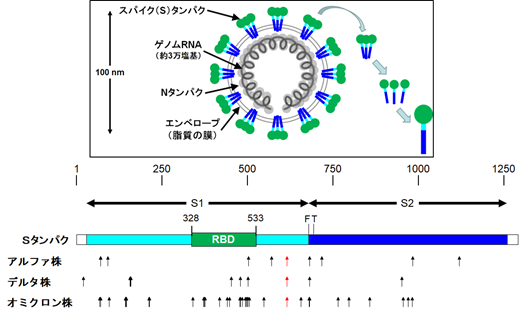
SARS-CoV-2の表面にはスパイク(S)タンパクが存在し【図1】、それがヒトの細胞表面にある受容体(ACE2というタンパク)と結合することによって感染が成立する。Sタンパクと受容体との関係は、いわば鍵と鍵穴のようなものであり、その相性は感染力の強弱に大きく影響する。一方、コウモリコロナウイルスRaTG13がSARS-CoV-2の祖先であると結論するには、一つ問題がある。Sタンパクの受容体結合領域の構造(つまり鍵の形)の類似性が低いのである(17アミノ酸のうち11アミノ酸のみが合致)。そんな中、Sタンパクの受容体結合領域に限って見ると、RaTG13よりもSARS-CoV-2とよく似たウイルスがセンザンコウから見つかった[10]。つまり、コウモリコロナウイルスが直接SARS-CoV-2の祖先になったのではなく、センザンコウコロナウイルスとの間でS遺伝子の組換えが起こってからヒトに伝播した可能性が浮かび上がったのである。
| 図1 |
| SARS-CoV-2のSタンパクの構造模式図と変異株に見られる変異部位 |
| 枠内は、SARS-CoV-2の構造模式図。Sタンパクはエンベロープ上で3量体として存在している。Sタンパク単量体はS1とS2から成り、S1には受容体結合部位(receptor-binding domain; RBD)が存在する。下の図は、Sタンパクを1次元的に表記したもので、フーリン切断部位(F)とTMPRSS2切断部位(T)が存在する。VOCのうち、アルファ株、デルタ株およびオミクロン株の変異部位がどこかが矢印(↑)で示してある。赤い矢印は、3者に共通のD614G変異に相当する。 |
 |
| 変異部位に関する図は、https://covariants.org/shared-mutations の情報に基づいて作成。 |
2種類のウイルスが同じ動物の同じ細胞に感染した場合、それぞれが複製する際にゲノムの組換えが起こって新たなウイルスを生ずるという現象は、コロナウイルスを含め、いろいろなウイルスで起こり得る。従って、センザンコウ経由説もあり得ないことではない。ちなみに、ゲノム配列の類似性に基づくSARS-CoV-2の国際的な系統分類として用いられるのがPhylogenetic Assignment of Named Global Outbreak Lineages(Pangolin)である[11, 12]。「センザンコウ」は英語で“pangolin”であることを考えると面白い命名である。
やはり「コウモリ」から?
最近になって、フランスのパスツール研究所のグループから興味深い報告があった[13]。本稿執筆時点では、まだ正式な論文としては発表されていないが、北ラオスのコウモリから見つかったコロナウイルス(BANAL-52)は、SARS-CoV-2との類似性(96.8%)がRaTG13に劣らないだけでなく、Sタンパクの受容体結合部位についても、17アミノ酸のうち16アミノ酸が合致していたというのである。しかも、BANAL-52のSタンパクは、SARS-CoV-2の受容体となるヒトのACE2に結合し、細胞内へのウイルスの侵入に寄与しうることが実験的に示された。従って、SARS-CoV-2はセンザンコウウイルスとコウモリコロナウイルスの組換えによって生まれたのではなく、BANAL-52のようなコウモリコロナウイルスが最も近縁の祖先になっている可能性が示唆されたことになる。ただし、このストーリーには2つの謎が残されている。
1つ目は、ラオスのコウモリコロナウイルスが、なぜ武漢を震源地とするパンデミックを引き起こしたのかということである。BANAL-52に感染したコウモリがラオスから武漢まで、1,500 km以上の距離を飛んで行ったのであろうか。ラトビアからスペインまでコウモリが2,000 km以上飛んだという記録もあるようなので[14]、あり得ないことではないかもしれないが、証明するのは難しいだろう。BANAL-52が種々の動物に(時には人間にも?)伝播しながら、SARS-CoV-2に姿を変えつつ武漢に向けて旅していったのであろうか。これは、2つの地点の間に生息するコウモリや他の動物にどのようなコロナウイルスが感染しているかを調べていけば明らかにできるかもしれない。あるいは、BANAL-52に感染した動物が人為的に武漢に運ばれてきて、ヒトに感染したのだろうか。これは、物流を調べれば分かるかもしれないが、野生動物の密猟や密輸が絡んでいると、表面化しないとも考えられる。いずれにしても、この謎の解明にはしばらく時間がかかりそうだ。
2つ目の謎は、SARS-CoV-2のSタンパクにはfurin(フーリン)という酵素による切断部位がある【図1】のに対し、BANAL-52には無いことである。コロナウイルスのSタンパクにはS1とS2という2つの領域があり、受容体への結合はS1が司る。S2は受容体に結合したウイルスの細胞内への侵入を司るが、その活性を発揮するには、SタンパクがS1とS2の間で切断されることが必須である。SARS-CoV-2の場合、まず標的細胞が産生するフーリンによって切断されることでS1とS2に開裂し、さらにTMPRSS2というタンパク切断酵素でS2の一部が切断されて効率よく機能を発揮するとされている【図1】。一方、BANAL-52のSタンパクにはフーリンによる切断部位が存在しないので、SARS-CoV-2への進化の途上でフーリン切断部位を獲得したことになる。他のウイルスとの組換えで獲得したのかもしれないが、その候補となるウイルスは見つかっていない。こちらの謎の解明も種々の野生動物のコロナウイルスに関する解析が必要であり、時間を要するかもしれない。
人工ウイルス説
武漢の研究所?
武漢には中国科学院のウイルス学研究所がある。1956年に武漢微生物学研究所として開設されたが、2002年のSARS発生以降、コロナウイルスの研究に注力している。研究所のある武漢がCOVID-19の震源地になったことから、SARS-CoV-2の起源については様々な憶測が流れた。その一つが、「SARS-CoV-2は、コウモリコロナウイルスを武漢の研究所で改変して作られた人工ウイルスだ」という説である。「患者検体の採取から10日足らずで30,000塩基もの全ゲノム情報を解読できたのは、もともとウイルスの存在を知っていたからではないか?」、「Sタンパクに存在するフーリン切断部位は、人為的に組み込まれたのではないか?」等の疑惑を指摘する声もあった。そして、「武漢ウイルス研究所のバイオセーフティ体制は脆弱であり、誰かが意図的に人工ウイルスを漏らしたのではないか」といった陰謀説も出てきた。今の所、これらを客観的に肯定する証拠も、否定する証拠も示されてはいない。ただ、たった10日間で原因ウイルスを同定し、その全ゲノム30,000塩基を解読できたのは、最近の中国の科学技術力に鑑みると、それほど不自然なことではないように思う。ちなみに現在は、COVID-19患者の検体を採取してからSARS-CoV-2の全ゲノムを解読するまで、日本でも早ければ2日以内で行える状況である。
中国は、研究所からのコロナウイルス漏出について苦い経験があるのは事実である。2002年に発生したSARSの流行は1年足らずで終息したが、北京の国立ウイルス学研究所では終息後もSARS-CoVの研究が続けられていた。2004年、その研究所で大学院生と研究員がそれぞれSARS-CoVに実験室内で感染し、それが市中感染に発展してしまった[15, 16]。死者も出て、600人もの接触者が隔離されることとなった。幸い、事態は迅速に収拾され、パンデミックに至ることはなかった。しかし、SRAS-CoV-2のように伝播力が強く、不顕性感染を起こしやすいウイルスであったら、違う顛末になっていたかもしれない。武漢の研究所からのウイルス漏出説が出てくるのは、こういった前例があることも理由の一つとなっている。人工ウイルスではないとしても、コウモリのコロナウイルスを研究していた研究者が実験室内で感染し、自覚の無いまま武漢市内を出歩いて、、、といったシナリオは否定できない。しかし、当事者でも見つからない限り、こういうことが起こったと証明するのは難しいだろう。逆に、起こらなかったという証明(いわゆる“悪魔の証明”)を求めるのも、無理筋のように思われる。
アメリカの関与?
SARS-CoV-2が武漢の研究所から漏出したとする説の急先鋒はアメリカであったが、それに対して中国政府はウイルスの「米国起源説」を主張して対抗した。逆ギレの言いがかりのように思えるが、必ずしもそれだけで片付けられない背景も存在する。コロナウイルスの研究は中国以外でも行われており、特にSARSの発生以降は、ヒトコロナウイルスの病原性や起源に関する研究が盛んになった。例えば米国では、SARS-CoVにコウモリコロナウイルスのSタンパク遺伝子を組み込んで、ヒトの細胞に感染する人工ウイルスを作製するような研究も行われていた[17]。ちなみにこれは武漢ウイルス学研究所との共同研究である。中国政府による「米国起源説」はこの研究を名指ししたものではないが、ヒトに対する感染力や病原性を有する人工コロナウイルスを作るのはどこでも可能であるというメッセージとして受け止めることができるかもしれない。
感染力や病原性が高まるかもしれない人工ウイルスを作ることは、現在の核酸合成技術や遺伝子組換え技術などを駆使すれば、それほど難しいことではない。このような研究(gain-of-function研究と呼ばれる)の意義やリスクについては、米国を中心に議論されてきた経緯がある[18]。2014年に、米国の国立研究機関である疾病予防管理センター(CDC)において高病原性インフルエンザウイルスと炭疽菌の誤発送や天然痘ウイルスの保管ミスが発覚するなど、バイオセキュリティ上の問題が露呈したこともあり、gain-of-function研究はそれまで以上に厳重な管理下に置かれることとなった。米国で行われた上記の人工コロナウイルス研究も、バイオセーフティーレベル3(BSL3)という厳重な封じ込め施設で行われている。そこからウイルスが漏れ出るといった事態は、余程のミスが重ならない限り起こり得ないと思われる。
新型コロナウイルスの変異
コロナウイルスはなぜ変異するのか?
ヒトのゲノム(遺伝情報)の本態は染色体のデオキシリボ核酸(DNA)であるのに対し、コロナウイルスのゲノムはリボ核酸(RNA)でできている。一般にRNAウイルスのゲノム複製は、RNAを鋳型としてRNA合成を行うRNA依存性RNAポリメラーゼ(RNA-dependent RNA polymerase; RdRp)が司る。コロナウイルスの30,000塩基のゲノムを複製するのは、原稿用紙75枚分の文章を書き写すようなものである。人間でも書き間違いをするように、コロナウイルスのRdRpも頻繁にミスをする。しかし、あまりミスが多いとウイルス増殖もうまくいかない。そこで、コロナウイルスはRdRpのミスを校正するnsp14という酵素を作ることができる[19]。いわば、誤字を修正するための消しゴムのような酵素であり、そのおかげで複製ミスは20分の1程度に減るとされるが、ミスがゼロになるわけではない。
コロナウイルスの変異頻度は、1回の複製あたり、ゲノムの各塩基について100万分の1程度の確率と推計されている[20]。コロナウイルスのゲノムの大きさは約30,000塩基であり、1回の複製で約1,000個の子孫ウイルスが作られるとすると、1個の親ウイルスが1サイクルの複製(約10時間)を経て産生する子孫ウイルス集団の中には、何らかの変異を持つウイルスが必ず含まれるという計算になる[20]。変異ウイルスのうち、増殖に不利な変異を持つものは淘汰されて消失するが、たまたま有利な変異を獲得したものは生き残り、優位に増殖していく。SARS-CoV-2は1か月あたり2塩基程度のスピードで変異しているとされるが、これは生き残った変異株、すなわち“勝ち組”ウイルスについて観測された数値である。
日本や海外でのウイルス変異の経緯
SARS-CoV-2は、武漢で最初に分離された株が標準株(A系統)として定義されている。日本では、2020年1月16日に武漢への渡航歴のある男性が最初のCOVID-19患者として見つかり[21]、その後しばらくの間、日本国内では武漢株の感染者が続いた(流行の第0波)[22]。2月に横浜に停泊したクルーズ船における集団感染事例で検出されたのも、武漢株とほぼ同一であった[22]。一方、2020年3月中旬に、ヨーロッパから流入したと考えられる株(欧州株;B.1.1.114系統)による流行(第1波)が始まり[22]、武漢株は見つからなくなっていった。武漢株はSタンパクの614番目のアミノ酸がアスパラギン酸であったのに対し、欧州株ではグリシンへと変異(D614G変異)しており、ウイルス表面に取り込まれるSタンパクの量が増えたり、Sタンパクの高次構造が変化したりすることで受容体との結合が促進され、増殖に有利になったと考えられている[23]。そして第1波が沈静化してきた6月に入ると、欧州株から6塩基変異した新たな株(B.1.1.284系統)が突然顕在化し、流行の第2波を生じた[24]。そして、10月からの流行第3波では、欧州株由来のさらに別系統(B.1.1.214系統)の変異ウイルスが主体となった[25]。これらの第2波と第3波の流行株に見られた変異は日本国内で起こったと考えられている。
海外に目を転ずると、英国では、2020年9月、SタンパクのD614G変異に加えて501番目のアミノ酸がアスパラギンからチロシンへと変異(N501Y変異)した新たな系統の変異ウイルスが検出された[26, 27]。アルファ株(B.1.1.7系統)と呼ばれているもので、それまでの流行株より伝播性が高く、英国国内で感染が拡大する一方、世界的にも拡散していった。日本最初のアルファ株は、2020年12月25日、空港検疫の際に検出された。その後、国内流入は止められず、流行第4波の原因となった。そして、2020年10月にインドで同定されたデルタ株(B.1.617.2系統)[27]は、伝播性がさらに高くなる変異や免疫が効きにくくなる変異を伴っており、“勝ち組”ウイルスとして世界中に蔓延していった。日本では2021年3月28日に空港検疫で検出されて以降、全国的に拡がり、各地で医療の逼迫を招くような大きな流行の波(第5波)を生ずることとなった。
アルファ株やデルタ株のように、伝播性の上昇や免疫の効果減弱などを伴う変異を生じた株はVOC(variant of concern; 懸念される変異株)と定義されている。例えば、基本再生産数(免疫の無い集団において1人の感染者から何人に感染が広がるか)を比較すると、標準株は2.5前後であったのに対し、アルファ株は4~5、デルタ株は5~8という推計がある[28]。インフルエンザウイルスは1~2、空気感染する水痘‐帯状疱疹ウイルスは8~10であることを考えると、デルタ株の伝播性の高さが分かる。また、国際的なデータベース(GISAID)に登録されたゲノム配列データ数に基づく推計では、アルファ株とデルタ株の伝播性は、標準株と比べてそれぞれ29%と97%上昇したとされている[29]。さらに、ワクチンによる免疫効果が減弱する可能性も報告されている。例えば、2021年7月、米国マサチューセッツ州(当時のワクチン接種率69%)で見つかった469名の感染者のうち、346名(74%)はワクチン接種を2回受けており、検出されたウイルスは約90%がデルタ株であった[30]。感染者のウイルス排出量についても、ワクチン接種の有無で有意な差が無かった[30]。つまり、現行のワクチンでは、デルタ株の“ブレークスルー感染”が容易に起こるし、感染源となるリスクも回避できないと考えられる。発症予防や重症化予防に関してはデルタ株にも有効とされるが、「自分はワクチンを接種したから感染源にはならない」という思い込みは禁物である。むしろ、感染しても自覚症状が出にくくなる分、気がつかないうちに他の人にうつすということの無いよう、注意が求められるかと思う。
SARS-CoV-2の変異で気になるのは病原性への影響である。現在検出されるSARS-CoV-2の殆ど全てに見られるD614G変異は、致死率や重症化、入院期間などに影響しないとされる[31]。ただし、SARS-CoV-2の見かけ上の病原性は、ウイルス自身の毒力だけでなく、感染者の総数や年齢分布、検査体制、医療体制、ワクチン接種を含む感染予防策の状況など、種々の因子が影響する。例えば、重症者が増えた場合、ウイルスの病原性が高まった可能性もあるが、それ以上に軽症者や無症状感染者が増えていれば、伝播性が高まったのであって、重症化率や病原性は低下したという見方もできる。英国ではデルタ株の蔓延時期に肺炎による入院患者の増加を認めたが、その一方で、鼻水、頭痛、咽頭痛など、一般的な風邪症状が自覚症状の上位を占めるようになったと報じられたこともある[32]。日本でも、デルタ株が主体となった流行第5波において、感染者数の増加に比べると重症者数や死者数は低く抑えられていた。ワクチンや医療体制の影響なども考えられるので、ウイルスの「弱毒化」という表現は軽々には使えないものの、変異に伴ってSARS-CoV-2の病原性も変わってきているように思われる。
一般に、ウイルスの変異はランダムに起こる現象である。そして、複製ミスによって生じた多様な変異ウイルスのうち、たまたま増殖に有利になった変異株が“勝ち組”として増え続ける。ウイルスにとって、ヒトに病気をおこすかどうかはある意味どうでもいいことで、効率良く伝播して増殖できるかどうかが重要である。例えば、感染者の肺から他の人の肺へと移動せねばならぬウイルスよりは、鼻から鼻への移動ですむウイルスの方が伝播性という点で有利である。また、多くの人に免疫ができれば、その免疫を回避できるウイルスの方が増殖に有利であり“勝ち組”となる。すなわち、ウイルスが進化する際、伝播性は高まり、免疫の効果は減弱するというのは珍しくないシナリオである。今までの所、SARS-CoV-2も同様のシナリオを辿って変異しているように見受けられる。新たな変異株が出現するたびに、「感染力が強そうだ!」、「ワクチンが効かないかもしれない!」といったことがメディアなどでは声高に報じられるが、自然の摂理に照らせば必然的な現象であり、驚くにあたらない。今後、治療薬が開発されて使われるようになれば、それが効かない耐性ウイルスの出現も想定の範囲内である。大切なのは、新たな変異株の病原性、特に重症化率や致死率への影響を迅速かつ客観的に評価できるかどうかということである。後述するように、本稿執筆中にオミクロン株という新たなVOCの出現が報じられたが[27]、これについても断片的な情報で一喜一憂するのではなく、病原性評価が重要になると考えられる。
新型コロナウイルスはどこに向かうのか
風邪コロナウイルスOC43から思うこと
風邪の原因はライノウイルスが最も多く(30~50%)、それに次いで多いのがコロナウイルス(10~15%)とされる[33]。現在までに、風邪の原因となるヒトコロナウイルス(HCoV)は4種類同定されており【表1】[34-37]、いずれも世界中で検出されている。
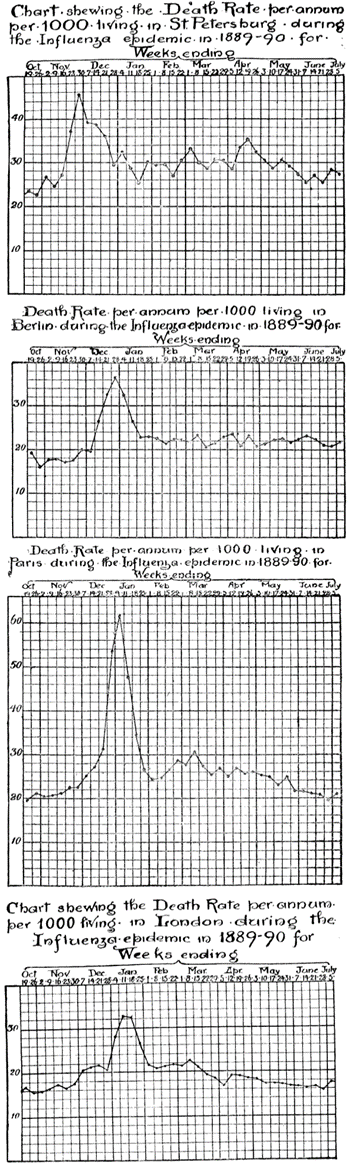
風邪の原因となるHCoVのうちOC43は1960年代に発見されたが、その起源はヒトに人獣共通感染を起こしたウシコロナウイルスではないかという論文が2005年に発表された[38]。ゲノム解析の結果から、ウシコロナウイルスがヒトに飛び火したのは1890年頃であったと推定され、これは「ロシアかぜ」のパンデミックが起こった時期(1889年~1891年)に重なる。ロシアかぜ発生当時はインフルエンザの流行と考えられており、英国人医師Richard Sisleyは 自身の著書にその頃の状況を記している[39]。1889年にロシア内陸部を震源として発生した肺炎が1890年にかけて西ヨーロッパに向けて拡がっていったようで、各地で超過死亡が1~2か月増加し、その流行地域がロシア→ドイツ→フランス→英国というように、東から西へと拡大している【図2】。その後、アメリカやアジアにも拡がり、日本には1890年4月頃に到来したとされる。
当時の日本の様子は、内務省衛生局が1922年にまとめたスペインかぜに関する報告書[40]の一部に記載されている。「各種ノ學校ハ患者夥クシテ其校ヲ閉ルノ仕合トナリ、健康上ハ勿論、教育上ニモ多少ノ損害ヲ與ヘタルハ掩フヘカラサルナリ。」というのは、COVID-19が学校教育に大きな影響を与えていることと重なる。ロシアかぜは小児や若い学生の患者が多かったことを意味するのかもしれない。当時はそもそも高齢者人口が少なく、高齢の患者が目立たなかったのかもしれないが、SARS-CoV-2のデルタ株やオミクロン株で若年層の感染者が多いことと符合しているようにも思われる。また、「大坂府下ニ於テ本年五月五日ヨリ去月二十四日マテニ届出タル流行性感冒患者總數ハ四千二十五人ニシテ爾後届出ノ患者ナク目下全ク終熄ニ歸セシモノノ如シ(同年八月十六日官報)」といった記述があり、大阪では3か月弱で終息したようである。日本での流行は1890年のうちに収まり、世界的にも1891年には終息したとされる。
| 図2 |
| ロシアかぜパンデミックの際の超過死亡の推移 |
| 1889年10月から1890年7月までの人口1,000人当たりの死者数を示したグラフ。上から順に、サンクトペテルブルク(ロシア)、ベルリン(ドイツ)、パリ(フランス)、ロンドン(イギリス)の様子を示している。死者数のピークは、それぞれ11月最終週、12月最終週、1月第1週、1月第2週と推移しており、ロシアからに西に向けてパンデミックが拡大している様子が見て取れる。 |
 |
| https://iiif.wellcomecollection.org/pdf/b20392175 |
ウイルスというものの存在すら知られておらず、当然PCRやワクチンも無かった時代に、当時の人類はコロナ禍(だったかもしれないもの)を2年で終息させたように見受けられる。そして、当初のウシコロナウイルスは変異を経て、風邪コロナウイルスOC43として人類との共存に落ち着いた…という可能性を、2005年の論文[38]は唱えている。すなわち、ロシアかぜの「終息」というのは、コロナ禍の「収束」であったとも考えられる。実は、他の風邪コロナウイルスも類似の動物コロナウイルスが見つかっており【表1】、「動物コロナウイルスの人獣共通感染 → 肺炎の流行 → ウイルスの変異 → 風邪コロナウイルスとして定着」というプロセスを人類は何度か経験している蓋然性は高い。「歴史は繰り返す」の言葉通り、SARS-CoV-2もまた、コウモリコロナウイルスがヒトに伝播したものであり、肺炎ウイルスから風邪ウイルスへの進化途上にあるのかもしれない。では、肺炎コロナウイルスと風邪コロナウイルスはどう区別すればいいのだろう。
風邪コロナウイルスHKU1から思うこと
風邪コロナウイルスの一つとされるHKU1【表1】は、2005年に香港で発見された(HKUはHong Kong Universityの略)[37]。実は、このウイルスは風邪の患者から分離されたのではなく、肺炎で2004年に入院した71歳の男性患者から見つかった[37]。遡って、2003年に肺炎で入院した35歳の女性患者(基礎疾患は特に無い)についても、保管してあった検体からHKU1が検出された[37]。つまりHKU1は、風邪コロナウイルスと称されるものの、入院を要するような肺炎を健常人にも引き起こすのである。また、香港からは、HKU1が検出された肺炎患者10人のうち2人(66歳の男性と72歳の男性)が亡くなったとの症例報告もある[41]。一方、SARS-CoV-2感染者の大多数は、軽症か無症状と言われる。従って、「HKU1は弱毒の風邪コロナウイルスで、SARS-CoV-2は強毒の肺炎ウイルス」という明瞭な線引きができるわけではなさそうである。
HKU1は香港だけに現局しているわけではない。米国オハイオ州からの報告では、呼吸器症状のある成人832人のうち13人からHKU1が検出され、そのうち7人は入院を要し、うち1人(75歳の男性)は敗血症様の症状(サイトカインストーム?)で亡くなっている[42]。日本でも患者検体からHKU1が検出されることは珍しくないし[43-47]、健常な小児から検出された例も報告されている[48]。
HKU1の発見は2005年であるが、これらの事実に照らすと、人類は今まで何十年も、あるいはそれ以上に亘って、このウイルスに曝されてきたと考えられる。従来、“原因不明の肺炎”で亡くなった患者の中には、HKU1が感染していた例もあったことと思う。ただ、殆どの人は気にかけなかったし、PCRで積極的に検出したり、ワクチンを作ろうとしたりということもなかった。もしかすると、誰も気にかけなかったがゆえに、多くの人が子供の頃に感染して重症化しないまま免疫を獲得し、「肺炎ウイルスの脅威」を「風邪ウイルスとの共存」に替えることができていたのかもしれない。実際、北京で行われた血清疫学調査の結果からは、多くの人が14歳までに4種類の風邪コロナウイルス全てに感染し、成人の7割以上が抗体を持っていることが示されている[49]。コロナウイルスの感染による免疫は終生免疫ではないので再感染も起こり得るが[50]、幼小児期の感染による免疫記憶のおかげで、その後に感染しても重症化を免れているのかもしれない。
オミクロン株の出現
2021年6月下旬以降、日本ではデルタ株の蔓延により流行の第5波を迎え、ピーク時には一日約26,000人もの新規感染者が報告された。日本の場合、COVID-19の患者が一人見つかると濃厚接触者を検査し、陽性者がいれば、さらにその人の濃厚接触者を検査するという具合に、症状の有無に関わらず、ネズミ算式に多くの人が検査対象となり、無症状感染者もたくさん見つかる。従って、COVID-19患者が少しでも増えると、報告される感染者総数は症状の有無にかかわらず加速度的に増加する。濃厚接触者の把握やPCR検査の段取りを担当する地域保健所の負担は極めて大きいようだ。一方、9月以降、新規感染者は急速に減少していった。その要因としては、ワクチンの効果による発症者の減少、“消しゴム”酵素nsp14の変異によるウイルスの過剰な変異、緊急事態宣言やマスク着用の効果など、いろいろ挙げられているが、それらが複合して関与している可能性もある。発症者が減少すると、検査対象となる濃厚接触者も加速度的に減少し、感染者数の激減に繋がったと思われる。本稿執筆時点では新規感染者が100人を切る日もあり、検査を受けていない無症状感染者の存在は否定できないものの、第5波は収束したと見做すことができそうである。
しかし、第5波収束の安堵感をかき消すように、新たな変異株の出現が報じられた。2021年11月10日前後に南アフリカ、ボツワナ、香港(南アからの渡航者)で行われたSARS-CoV-2のゲノム解析結果から、多数の変異を有する新しい変異株の存在が明らかとなった。11月24日に南アフリカからWHOに報告されたが、Sタンパクに見られる変異の部位や数から【図1】、それまで蔓延していたデルタ株(B.1.617.2系統)とは独立に生じた変異株と考えられ、オミクロン株(B.1.1.529系統)と命名された[27]。そして、WHOは11月26日に[27]、日本の国立感染症研究所は11月28日に[51]、オミクロン株をVOCと位置付けた。アルファ、ベータ、ガンマ、デルタに次いで、5つ目のVOCということになる。南アフリカでのウイルスゲノム解析結果によると、検出されたSARS-CoV-2の7割以上をオミクロン株が占めており[52]、デルタ株を凌駕する高い伝播性を有すると思われる。また、ワクチン接種者の抗体はオミクロン株に対する効果が低かったり[53]、既感染者へのオミクロン株の再感染が頻繁に見られたりする[54]など、免疫効果の減弱も疑われている[55]。オミクロン株が南アフリカで生じたものなのか、他国で誕生して南アフリカに流入したのかについては、定かでない。
一方、オミクロン株は重症化しにくいかもしれないとの情報がWHOのアフリカ地域事務局からは出てきている[56]。南アフリカでの入院者数や集中治療室の使用率はデルタ株の流行時に比べると低く抑えられているという。見つかった感染者の多くが若年者であるため重症例が少ないとの見方もあるが、オミクロン株自体が弱毒化(風邪ウイルス化)している可能性もある。オミクロン株の病原性を正確に評価するためにはまだ時間を要するが、コウモリ由来の肺炎ウイルスからヒトの風邪コロナウイルスへとさらに一歩近づいたのであれば、新たな変異株の出現は必ずしも悪いニュースとは限らない。興味深いことに、オミクロン株はSARS-CoV-2と風邪コロナウイルスとの間での組換えが(おそらく重複感染したヒトの細胞内で)起こって生じた可能性を示唆する論文も(現時点では査読未完了であるが)出てきている[57]。
おわりに
SARS-CoV-2は軽症者や無症状感染者の比率が高いため、根絶することは困難であると当初から思われていた。また、風邪コロナウイルスHKU1でも肺炎の症例や死亡例があるように、SARS-CoV-2が弱毒化しても、重症者や死者をゼロにすることは不可能だろう。従って、変異ゼロや死者ゼロを待っていては、いつまで経ってもパンデミックは終わらず、日常生活の不自由さや経済へのダメージも解消されない。新たな変異株が見つかったと聞くと人々は不安になり、時に社会の混乱も生ずるが、変異が無ければ弱毒化も起こらないのである。「ウイルスとは変異するものである」という自然の摂理と正面から向き合い、変異株の性質(特に病原性)を冷静かつ客観的に評価することが必要ではないかと思う。
SARS-CoV-2やその変異株が肺炎ウイルスか風邪ウイルスか、パンデミックは収束したか否かという線引きは、あくまで人間の判断で行うことである。コロナ禍が長引くことを望む人はいないと思うが、ポリティクスやコマーシャリズムの影響で恣意的な線引きがなされたり、判断の時期が必要以上に先延ばしされたりすることは望ましくない。思惑や駆け引きに左右されず、バイアスを排除した科学的根拠に立脚して判断することができれば、コロナ禍の国際的な出口戦略も見えてくるかと思われる。その際、ワクチンや治療薬の開発や利用は有意義かもしれない。ただし、デルタ株やオミクロン株がそうであるように、ワクチン効果を減弱させる変異株、あるいはインフルエンザウイルスなどで見られたように、薬の効かない耐性株が出現することは想定しておく必要がある。また、SARS-CoV-2が風邪ウイルス化したとしても重症患者がゼロにはならないことを前提として、感染症全般に関する医療提供体制を整備しておくことは肝要である。
ウイルス感染によってヒトが病気になったり、命を落したりするのは困ったことである。しかし、ヒトを含めて種々の生物は、それらを宿主とする様々なウイルスとの共進化を遂げてきた。そもそも我々の染色体にはウイルス遺伝子の遺残物(内在性ウイルス)が存在するし、免疫機構も人類の祖先がいろいろなウイルスに曝されたことで進化してきたと思われる。従って、「ウイルス感染=絶対悪」という図式は、生物の進化と言う俯瞰的な視点からは必ずしも成り立たない。一方、新たなウイルスが動物からヒトに飛び火してパンデミックを引き起こす懸念は、今後も払拭されない。その時に人類がどう振る舞い、どう対応するのがベストなのかを考える上で、COVID-19やSARS-CoV-2は現在進行形の教材である。多くの人の健康や命が失われたことは悲劇であり、末席ウイルス学者として無力さを悔いるところ大であるが、次代を担う若者たちがこの教材を最大限に活用してくれることを心より願っている。
[引用文献]
(査読が完了していない論文や予備的知見に関するネット情報を含むので、ご留意いただきたい。)
- N Engl J Med. 2020 Feb 20;382(8):727-733.
- https://www.who.int/news/item/27-04-2020-who-timeline—covid-19
- Nature. 2020 Mar;579(7798):265-269.
- N Engl J Med. 2003 May 15;348(20):1967-76.
- Proc Natl Acad Sci U S A. 2005 Sep 27;102(39):14040-5.
- Science. 2003 Oct 10;302(5643):276-8.
- Science. 2005 Oct 28;310(5748):676-9.
- N Engl J Med. 2012 Nov 8;367(19):1814-20.
- Nature. 2020 Mar;579(7798):270-273.
- Nature. 2020 Jul;583(7815):282-285.
- Nat Microbiol. 2020 Nov;5(11):1403-1407.
- https://cov-lineages.org/
- https://www.researchsquare.com/article/rs-871965/v1
- Mammalia. 2020 Oct 21;85(2):161–163.
- https://www.who.int/emergencies/disease-outbreak-news/item/2004_04_30-en
- Science. 2004 Apr 30;304(5671):659-61.
- Nat Med. 2015 Dec;21(12):1508-13.
- https://www.washingtonpost.com/nation/interactive/2021/a-science-in-the-shadows/
- Proc Natl Acad Sci U S A. 2006 Mar 28;103(13):5108-13.
- Elife. 2020 Apr 2;9:e57309.
- https://www.mhlw.go.jp/stf/newpage_08906.html
- https://www.niid.go.jp/niid/ja/basic-science/467-genome/9586-genome-2020-1.html
- Biochem Biophys Res Commun. 2021 Jan 29;538:108-115.
- https://www.niid.go.jp/niid/ja/basic-science/467-genome/9787-genome-2020-2.html
- https://www.niid.go.jp/niid/ja/diseases/ka/coronavirus/2019-ncov/2488-idsc/iasr-news/10152-493p01.html
- https://virological.org/t/preliminary-genomic-characterisation-of-an-emergent-sars-cov-2-lineage-in-the-uk-defined-by-a-novel-set-of-spike-mutations/563
- https://www.who.int/en/activities/tracking-SARS-CoV-2-variants
- https://newsroom.unsw.edu.au/news/health/what-we-know-about-sars-cov-2-delta-variant
- Euro Surveill. 2021 Jun;26(24):2100509.
- MMWR Morb Mortal Wkly Rep. 2021 Aug 6;70(31):1059-1062.
- J Clin Med. 2021 Jun 15;10(12):2635.
- https://www.bbc.com/news/health-57467051
- Lancet. 2003 Jan 4;361(9351):51-9.
- Proc Soc Exp Biol Med. 1966 Jan;121(1):190-3.
- Proc Natl Acad Sci U S A. 1967 Dec;58(6):2268-73.
- Nat Med. 2004 Apr;10(4):368-73.
- J Virol. 2005 Jan;79(2):884-95.
- J Virol. 2005 Feb;79(3):1595-604.
- Sisley R. Epidemic Influenza: Notes on Its Origin and Method of Spread. London: Longmans, Green, and Co.; 1891. https://iiif.wellcomecollection.org/pdf/b20392175
- 内務省衛生局.流行性感冒.第二章 我邦ニ於ケル既往ノ流行槪況.1922.
https://www.niph.go.jp/toshokan/koten/Statistics/PDF/100088820002.pdf - J Infect Dis. 2005 Dec 1;192(11):1898-907.
- Open Forum Infect Dis. 2017 Mar 25;4(2):ofx052.
- Jpn J Infect Dis. 2015;68(2):138-41.
- Jpn J Infect Dis. 2015;68(6):523-5.
- BMC Infect Dis. 2020 Jun 1;20(1):388.
- Jpn J Infect Dis. 2020 Sep 24;73(5):394-397.
- Jpn J Infect Dis. 2021 Aug 31.doi: 10.7883/yoken.JJID.2021.250. Online ahead of print.
- J Clin Virol. 2015 Mar;64:59-63.
- BMC Infect Dis. 2013 Sep 16;13:433.
- Epidemiol Infect. 1990 Oct;105(2):435-46.
- https://www.niid.go.jp/niid/ja/2019-ncov/2551-cepr/10792-cepr-b11529-2.html
- https://www.nicd.ac.za/wp-content/uploads/2021/12/Update-of-SA-sequencing-data-from-GISAID-8-Dec-21_Final.pdf
- https://www.medrxiv.org/content/10.1101/2021.12.07.21267432v1.full.pdf
- https://www.medrxiv.org/content/10.1101/2021.11.11.21266068v2
- https://www.nature.com/articles/d41586-021-03672-3
- https://www.afro.who.int/news/omicron-spreads-severe-cases-remain-low-south-africa
- https://osf.io/f7txy/