注:この記事は、有識者個人の意見です。COVID-19有識者会議の見解ではないことに留意ください。
- ウイルス感染症は、ウイルスと宿主の相互作用の結果であり、双方の性質を理解するとともに両者の相互作用を理解することが重要である。
- 新型コロナウイルス感染症(COVID-19)では、一般的なウイルス感染症と同様に、感染への初動対応であるインターフェロン応答を中心とする自然免疫と、引き続いて起こる抗体・CTLによる獲得免疫が誘導される。
- COVID-19重症者では、感染直後のインターフェロン応答が適切に作用していないことを示す知見が蓄積されている。
- 一方、原因ウイルスであるSARS-CoV-2は宿主免疫応答を回避する機構を複数有していることが明らかとなっている。
- SARS-CoV-2特有のウイルス―宿主相互作用の結果、COVID-19の病態が形成されていると考えられるが、そのメカニズム解明にはさらなる科学的知見の蓄積が必要である。
はじめに
新型コロナウイルス(SARS-CoV-2)の出現から、はや2年が経とうとしている。その間、新型コロナウイルス感染症(COVID-19)に対する疫学的・臨床的・基礎医学的知見が急速に蓄積されている。しかしながら、SARS-CoV-2のウイルス学的性質、感染に対する宿主応答については未だ理解は不十分である。ウイルスが宿主(ヒトや動物など)体内に侵入し、宿主細胞に感染することで感染が成立するが、ウイルス感染そのものが必ずしも病気を起こす(=感染症の発症)わけではない。感染症では、免疫応答を主体とする宿主反応がウイルスを排除しようとすることにより、さまざまな症状を示す。一方で、ウイルスは宿主反応に対抗して排除を逃れ、子孫ウイルスを増やし、他者へ感染することでウイルス自身を存続させようとする。ウイルスと宿主が互いに作用し合った結果がウイルス感染症であるため、その双方の性質を理解するとともに、両者の相互作用を理解することが重要である。本稿では、これまでに明らかになっている知見を整理し、COVID-19におけるウイルス―宿主間相互作用について考えてみたい。
ウイルス感染に対する免疫応答
自然免疫によるウイルスの侵入感知から、獲得免疫の発動へ
私たちの免疫システムは、大きく分けて自然免疫応答と獲得免疫応答に分けられる。自然免疫応答は、ウイルスなどの外敵侵入を感知し、初動対応として発熱等に代表される炎症反応やインターフェロンなど抗ウイルス作用のある分子の産生を惹起することで外敵の排除を行う。その一方で、さらに高度な免疫システムへ外敵侵入を知らせる警報の役目も担っている。自然免疫応答は、それぞれのウイルスに特異的に働く「抗体」と「細胞傷害性Tリンパ球(CTL)」による獲得免疫応答を開始させるために必須である。自然免疫・獲得免疫両者が正常に機能することでウイルスの排除やコントロールが可能となる。自然免疫開始から獲得免疫応答誘導に至る一連の過程は、数多くの免疫担当細胞が関与する非常に複雑なシステムの上に成り立っている。各免疫反応は”適切な時期”に”適切な程度“で機能することが重要であり、不適切な過度の免疫反応は組織障害やサイトカインストームなどを引き起こす”諸刃の刃”となる。
SARS-CoV-2感染におけるウイルス―宿主間相互作用
インターフェロン応答を中心とした自然免疫応答
複数の種類からなるインターフェロン(IFNs)は、その名の由来の通り、ウイルス感染を抑制する機能を持つサイトカインであり、自然免疫応答の中心的役割を担う。通常、ウイルス感染が起きると、感染局所でウイルスを感知した細胞が迅速にインターフェロンを産生、局所の細胞でISG (Interferon Stimulated Gene)と呼ばれるインターフェロンによって発現が誘導される遺伝子群が発現し、ウイルス感染の拡がりが抑制される。COVID-19患者の解析では、感染初期にはほぼ全ての患者で血中のインターフェロンα、IL-1βなど炎症性サイトカインの上昇が見られ、軽症者では一定期間で減少に転じるのに対して、重症者では経過を通して上昇し続けており、入院期間や致死率と関連していた[4]。また別の解析では、感染初期の血中のインターフェロンα2量・活性が、重症者では軽症/中等症患者に比して低いことが示されており[5]、インターフェロン応答は感染からの時期や患者間で大きく異なることが明らかとなっている。
一方、ゲノム解析ではインターフェロン産生に関連する遺伝子多型が重症化リスクと関連することが示されており[1]、重症者の10%でI型インターフェロンに対する自己抗体を保有していたのに対して、軽症/無症状者では皆無であったことが報告されている[2]。また、動物実験ではインターフェロン受容体欠損マウスでは野生型マウスに比べてウイルス複製増加、肺機能低下が確認されており[3]、COVID-19におけるインターフェロン応答の重要性は明らかになりつつある。そのため、インターフェロンについては、COIVD-19に対する治療の可能性が検討されている。動物実験では、予防的あるいは感染時でのインターフェロン投与はSARS-CoV-2の感染や重症化を阻止する効果が見られるのに対し[6]、発症後の投与では効果がなかったことから[7]、感染直後のインターフェロン応答はCOVID-19において防御的に働くことが示唆される。インターフェロン投与の臨床試験も複数行われており、その臨床効果は未だ一定の評価が得られていないが、感染初期に投与した場合に一定の効果が見られているようである[8][9]。
重症COVID-19では、感染初期でのインターフェロン応答の不足あるいは遅延が原因となり、体内で増殖したウイルスに対する反応として、長期間にわたるインターフェロン応答等炎症性サイトカイン産生が持続し、結果として重症化を引き起こしている可能性が考えられる。
獲得免疫応答
獲得免疫応答で外敵排除に働くのが、抗体とCTLである【図1】。抗体はウイルス粒子表面のタンパク質に結合し、細胞への侵入に必要な受容体との結合を阻害することでウイルスを中和し、細胞への侵入を阻止する。一方、CTLは、ウイルス感染細胞上でヒト白血球抗原(HLA)と呼ばれる分子に提示された抗原=ウイルスタンパク質のかけら(エピトープ)を認識し、ウイルス感染細胞を破壊することでウイルス感染を抑制する。例えれば、抗体は個々の戦闘機(ウイルス)の攻撃力を失わせるのに対して、CTLは戦闘機の発着場である母艦(ウイルス感染細胞)を攻撃・破壊しているようなものであり、両者が機能することで体内からのウイルス排除を行う。
| 図1 |
| 獲得免疫応答での抗原認識 |
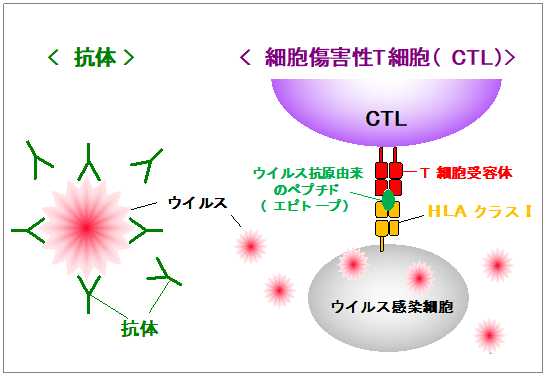 |
抗体はB細胞により産生されるが、ナイーブB細胞が抗原(SARS-CoV-2のウイルスタンパク質)と出会うことで抗体を産生する抗体産生細胞となる。また、ナイーブCD8陽性T細胞がウイルス由来エピトープを認識し、機能を獲得することでCTLとなる。ナイーブB細胞・CD8陽性T細胞が抗体産生能・細胞傷害能を獲得するためには、いずれもCD4陽性T細胞による助け(ヘルパー機能)が必要で、CD4陽性T細胞がヘルパー機能を発揮するためには自然免疫応答が誘導されていることが前提となる。
多くのウイルス感染では、通常IgMが先行して出現し、その後IgGやIgAへとクラススイッチが起こる。SARS-CoV-2感染では、発症後2週間以内にIgM、IgAとほぼ同時にS、Nに対するIgG抗体が出現し、95%以上の感染者で血清抗体陽転化する[10][11]。SARS-CoV-2に対する血中抗体量は重症度と比例しており、重症者で最も高く、質的指標である中和能や抗体の多様性も重症者の方が高い[12][13]。高い抗体量・体内進化によって得られる高中和能・多様な抗体産生は抗原量(=ウイルス量)に依存すること、軽症者に比して重症者では鼻咽頭でのウイルス量が高く、ウイルス排除までの時間が長いことを考え合わせると、重症者で見られる強い抗体応答は、高い抗原量に対して反応した”結果”であり、必ずしもウイルスコントロールに寄与していないとも解釈できる。
現在、SARS-CoV-2に対する2種類の中和抗体のカクテル療法は、数少ないCOVID-19に対する治療法として使用されている。発症から時間の経っていない軽症者を対象として重症化阻止に治療効果が確認されているが、自身の免疫応答によるSARS-CoV-2抗体が出現する前(あるいは低値である段階)に抗体を供給し、ウイルス量を低減させることで、上述したような自然免疫破綻を阻止していると考えられる。
一方、SARS-CoV-2特異的T細胞については、末梢血中では抗体よりも早く検出され、ほぼ全ての感染者で2週間以内に検出される[14]。感染初期のCOVID-19患者検体の解析では、CD4陽性T細胞応答が重症化の阻止と関連が見られており、軽症者では早期にCD4陽性T細胞応答が見られるのに対して、CD4陽性T細胞応答の不在が重症化や死亡と関連が見られている[15]。一方、CD8陽性T細胞応答についても、CD4陽性T細胞よりも頻度は低いが、軽症状と関連が見られている[16]。PerforinやGranzymeといった機能分子の発現能を有しており、感染者体内でSARS-CoV-2感染細胞を破壊するCTLとして機能していることが示唆されている。
CD4陽性T細胞は、上述したようにB細胞やCD8陽性T細胞の機能発現に必須であるのみでなく、さまざまな免疫細胞を局所にリクルートし、自身も抗ウイルス作用を有する。SARS-CoV-2特異的なCD4およびCD8陽性T細胞が、感染初期の早い段階で適切に誘導されることが、重症化阻止に貢献しているかもしれない。T細胞応答の解析には文字通り”細胞”が必要となるため、血漿のみで解析可能な抗体に比べて検体収集や実験上の制約が多く、抗体に比べて研究が遅れているのが現状である。また、上述したように、T細胞応答はHLAに依存する。HLA遺伝子は、ヒトゲノムで最も多様性に富む遺伝子で、人種によってその遺伝子型は大きく異なる。T細胞応答については、各地域での、臨床検体を用いた、より詳細な解析を進めることが重要である。
新型コロナウイルスの宿主免疫応答からの回避戦略
ここまで、SARS-CoV-2感染に対する宿主側の反応について説明した。一方、ウイルス感染に対する宿主防御機能としての免疫応答に対して、さまざまなウイルスが自身の生存のために宿主免疫応答を回避する独自の戦略を有していることが知られている。
自然免疫からの回避:
自然免疫応答はウイルス感染に対する防御機構として最初に機能するものである。ウイルスに対する免疫応答は、細胞に存在するパターン認識受容体(Pattern Recognition Receptors, PRR)と呼ばれる分子にウイルス由来の核酸が結合することで開始される。コロナウイルスは複製の際、細胞質内で二重膜小胞を形成し、その内部でゲノムRNAが複製される。このような複製様式を取ることにより、PRRからウイルスゲノムを物理的に隔離することで、その認識から逃れている可能性が考えられる。また、コロナウイルスは、ウイルス複製に必須ではない多くのアクセサリータンパク質を有しているが、一部は免疫回避や病態形成に寄与していると考えられており、その中の多くが、インターフェロン応答に拮抗するあるいはインターフェロン応答から逃れる機能を有することが知られている[17]。これらのウイルス側の戦略は、インターフェロン産生に至るさまざまな過程を阻害することにより免疫応答を減弱/遅延させ、宿主個体での感染を成立させようとしていると考えられる。
獲得免疫からの回避:
獲得免疫からの回避機構も、さまざまなウイルスで備わっている。HSV、CMV、など多くのヘルペスウイルスやHIVなどは、HLAによるT細胞への抗原提示を低下させるウイルスタンパク質を有しており、CTLによる認識から逃れているが、SARS-CoV-2も同様の免疫回避機構を有することが明らかとなっている。SARS-CoV-2とSARS-CoVは、ゲノム全体では79%という高い相同性を示すが、ORF8と呼ばれる遺伝子のみ塩基配列の相同性が20%程度と低く、その機能も異なることが予想されていた。SARS-CoV-2のORF8は直接HLA分子と結合し、HLA分子をオートファジーを介したリソソーム分解の標的とすることでHLAによる抗原提示を低下させていることが明らかとなっている(SARS-CoVのORF8はこの機能を有していない)[18]。
これらの宿主免疫回避機構は、ウイルスが意図的に、あらかじめこれらの機能を備えていた、というよりは、ウイルスの進化の過程で、免疫回避機構を獲得したウイルスが選択されてきたと考える方が自然である。コロナウイルスは、古くから地球上に広く存在し、多様な動物を宿主とするウイルス種である。長い歴史の中で、種の存続に有利な様々な戦略を獲得してきたのではないだろうか。
SARS-CoV-2感染におけるウイルスと宿主相互作用について、以下のようなモデルが考えられる【図2】。
| 図2 |
| SARS-CoV-2感染における宿主免疫応答モデル |
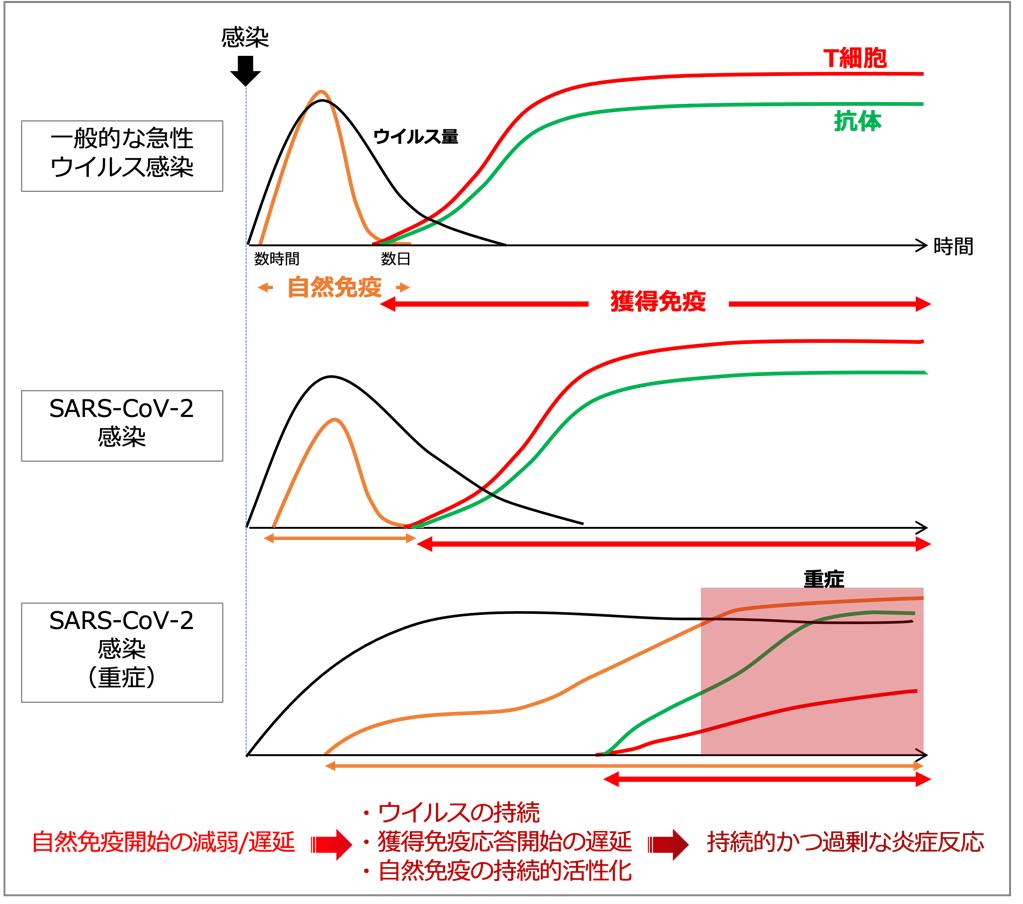 |
一般的なウイルス急性感染に対する免疫応答では、ウイルス感染後速やかに自然免疫が誘導され、数時間でインターフェロンや炎症性サイトカイン産生を開始、局所でのウイルス減少とともに自然免疫応答は沈静化、引き続き獲得免疫応答が適切なタイミングで誘導され、ウイルスを消失させる。軽症・中等症程度のSARS-CoV-2感染では、ウイルスの自然免疫回避機構によりインターフェロン応答など自然免疫誘導が減弱あるいは遅延するが、自然免疫によるインターフェロン/炎症性サイトカイン産生は一過性に推移、獲得免疫応答も誘導され、ウイルスを排除し治癒に至る。一方、重症者では、ウイルス側あるいは宿主側の要因で感染初期に自然免疫応答が適切に誘導されず、獲得免疫の出現も遅れた結果、高ウイルス量が持続し、抗原量依存性に免疫系は長期間活性化、持続的な高炎症状態(暴走状態)に陥る。感染局所である肺には活性化した免疫細胞が集積し、炎症性サイトカインが過剰に産生され(サイトカインストーム)、急性呼吸窮迫症候群(ARDS)を発症する。
これらのモデルは、あくまで現時点で得られている科学的知見、主にCOVID-19患者の末梢血検体を使用した解析から得られた知見を基としている。COVID-19での”感染局所”は肺などの呼吸器であるため、末梢血中で観察される現象は必ずしも局所の病態を反映しているとは言えないことに留意する必要がある。COVID-19の重症化(組織傷害や過剰炎症・サイトカインストームによるARDS等)にSARS-CoV-2特異的T細胞・抗体応答が加担している可能性も考えられており、その真偽を明らかにするにはさらなる科学的検証が必要である。COVID-19の免疫病態を正しく理解するためには、さらなる臨床検体を用いての解析、病理検体や動物モデル等を用いた肺組織での詳細な免疫学的解析が重要である。
ウイルス―宿主間相互作用と変異ウイルスの出現
上記とは異なる免疫回避機構としてウイルス生存戦略に貢献しているのが、ウイルス変異である。ウイルスの変異は、ゲノム複製の際に一定の確率で起こる複製エラーによる。RNAウイルスは、DNAウイルスと比較してエラー率が高く、変異を起こしやすい。一般的に、複製エラーはゲノム上のあらゆる場所でランダムに起こると考えられるが、たまたま抗体の結合部位や、エピトープとしてHLAに結合する部位にアミノ酸置換を伴う変異が生ずると、抗体やCTLの認識から逃れることがあり、このような獲得免疫から逃避した変異ウイルスはその個体で選択的に増殖することが可能となる。HIVは非常に変異を起こしやすいウイルスであり、CTLや抗体からの逃避変異ウイルスが容易に出現することが知られている。一方、SARS-CoV-2では複製の際のエラー率は他のRNAウイルスに比して低いと考えられており、実際にウイルス間で遺伝的多様性を比較すると、コロナウイルスはHIV、HCV、インフルエンザウイルスなどの他RNAウイルスと比べると、格段に低い[19]。しかしながら、現在のSARS-CoV-2の全世界での急速な感染拡大においては、世界中で猛烈な勢いで日々ウイルス複製が行なわれているため、変異ウイルス出現の機会は高いと考えられる。実際に、既に世界中で変異ウイルスの出現が問題となっている。Variant of Concern (VOC)とされているベータ株(B.1.351)、ガンマ株(P.1)は、Sタンパク質の受容体結合部位(RBD)に位置する484番目のアミノ酸にE484Kと呼ばれるアミノ酸変異を持つが、この変異を有するウイルスは中和抗体に抵抗性を持つことが実験的に確認されおり[20]、中和抗体からの逃避変異ウイルスとして選択された可能性が考えられる。一方、アルファ・ベータ・ガンマ株が共有するN501Y変異はSタンパク質と宿主細胞の受容体であるACE2の結合力が高めることを示唆するデータが得られており[21]、その結果感染性が増強し選択されてきたと考えられることができる。
現在、日本の流行株の大部分を占めているデルタ株(B.1.617.2)はRBDにL452RとT478Kという変異を有しており、中和抗体の中和活性が野生型と比べて低いことが示されている[22]。また、L452R変異は、日本人の多くが保有するHLA-A*24:02に提示されるCTLのエピトープに含まれており、この変異を有するウイルスはCTL認識から逃れることが報告されている[23]。
ここまで書くと、一方的にウイルスが宿主免疫から逃れているように思われるが、宿主免疫応答は、出現した変異ウイルスに対して効果的に働く抗体やT細胞を、新たに誘導することも知られている。さらに、T細胞への抗原提示分子であるHLAに提示されるエピトープは【図1】、SARS-CoV-2ウイルスタンパク質のほぼ全体に分布しており、既に1000種類を超えるエピトープが報告されている[24]。実際に、COVID-19回復者の末梢血中には30-40種類の異なるエピトープを認識するSARS-CoV-2特異的T細胞が検出されており[25]、1箇所のアミノ酸置換による逃避変異ウイルスの出現が、免疫学的破綻に直結することは考えにくい。しかしながら、ウイルスは一定の選択圧の中で複製を繰り返すことで、免疫からの逃避変異や感染増強に寄与する変異を獲得する機会を得る。このような”ウイルスに有利な変異”が蓄積していくことで、病原性の高いウイルスが出現する危険性が高まる。実際に、私たちはHIV感染症において、日本人集団では日本人の半数以上が有するHLA-A*24:02に拘束されるCTLからの逃避変異ウイルスが流行していることを報告している[26]。ウイルス感染症において変異ウイルスの出現を抑えるには、ウイルスに複製の機会を与えないこと、すなわちワクチン接種をはじめとする感染予防対策により、全世界で”感染”というイベントを極力抑えこむ努力が最も重要であると考える。
おわりに
本稿では、COVID-19でのウイルスと対する宿主反応としての免疫応答について、これまでに明らかとなっている知見についてまとめたが、この感染症の本質については、依然未解明である。全世界で脅威的な速さでさまざまな研究が進められているが、引き続き科学的知見を蓄積していくことが重要である。
今後、このウイルスがどのように変化(進化)していくのか、感染性や病原性がどのように変わっていくのか、感染拡大がどのように推移していくのか、その挙動を予測することは、現時点では不可能である。明らかなことは、今後の新型コロナウイルスの挙動は、宿主である私たちの挙動に依存しているということである。
[引用文献]
- Zhang, Q. et al. Inborn errors of type I IFN immunity in patients with life-threatening COVID-19. Science 370, doi:10.1126/science.abd4570 (2020).
- Bastard, P. et al. Autoantibodies against type I IFNs in patients with life-threatening COVID-19. Science 370, doi:10.1126/science.abd4585 (2020).
- Leist, S. R. et al. A Mouse-Adapted SARS-CoV-2 Induces Acute Lung Injury and Mortality in Standard Laboratory Mice. Cell 183, 1070-1085 e1012, doi:10.1016/j.cell.2020.09.050 (2020).
- Lucas, C. et al. Longitudinal analyses reveal immunological misfiring in severe COVID-19. Nature 584, 463-469, doi:10.1038/s41586-020-2588-y (2020).
- Hadjadj, J. et al. Impaired type I interferon activity and inflammatory responses in severe COVID-19 patients. Science 369, 718-724, doi:10.1126/science.abc6027 (2020).
- Hoagland, D. A. et al. Leveraging the antiviral type I interferon system as a first line of defense against SARS-CoV-2 pathogenicity. Immunity 54, 557-570 e555, doi:10.1016/j.immuni.2021.01.017 (2021).
- Bessiere, P. et al. Intranasal type I interferon treatment is beneficial only when administered before clinical signs onset in the SARS-CoV-2 hamster model. PLoS Pathog 17, e1009427, doi:10.1371/journal.ppat.1009427 (2021).
- Wang, N. et al. Retrospective Multicenter Cohort Study Shows Early Interferon Therapy Is Associated with Favorable Clinical Responses in COVID-19 Patients. Cell Host Microbe 28, 455-464 e452, doi:10.1016/j.chom.2020.07.005 (2020).
- Zhou, Q. et al. Interferon-alpha2b Treatment for COVID-19 Is Associated with Improvements in Lung Abnormalities. Viruses 13, doi:10.3390/v13010044 (2020).
- Roltgen, K. et al. Defining the features and duration of antibody responses to SARS-CoV-2 infection associated with disease severity and outcome. Sci Immunol 5, doi:10.1126/sciimmunol.abe0240 (2020).
- Wajnberg, A. et al. Robust neutralizing antibodies to SARS-CoV-2 infection persist for months. Science 370, 1227-1230, doi:10.1126/science.abd7728 (2020).
- Piccoli, L. et al. Mapping Neutralizing and Immunodominant Sites on the SARS-CoV-2 Spike Receptor-Binding Domain by Structure-Guided High-Resolution Serology. Cell 183, 1024-1042 e1021, doi:10.1016/j.cell.2020.09.037 (2020).
- Shrock, E. et al. Viral epitope profiling of COVID-19 patients reveals cross-reactivity and correlates of severity. Science 370, doi:10.1126/science.abd4250 (2020).
- Grifoni, A. et al. Targets of T Cell Responses to SARS-CoV-2 Coronavirus in Humans with COVID-19 Disease and Unexposed Individuals. Cell 181, 1489-1501 e1415, doi:10.1016/j.cell.2020.05.015 (2020).
- Tan, A. T. et al. Early induction of functional SARS-CoV-2-specific T cells associates with rapid viral clearance and mild disease in COVID-19 patients. Cell Rep 34, 108728, doi:10.1016/j.celrep.2021.108728 (2021).
- Rydyznski Moderbacher, C. et al. Antigen-Specific Adaptive Immunity to SARS-CoV-2 in Acute COVID-19 and Associations with Age and Disease Severity. Cell 183, 996-1012 e1019, doi:10.1016/j.cell.2020.09.038 (2020).
- Lowery, S. A., Sariol, A. & Perlman, S. Innate immune and inflammatory responses to SARS-CoV-2: Implications for COVID-19. Cell Host Microbe 29, 1052-1062, doi:10.1016/j.chom.2021.05.004 (2021).
- Zhang, Y. et al. The ORF8 protein of SARS-CoV-2 mediates immune evasion through down-regulating MHC-Iota. Proc Natl Acad Sci U S A 118, doi:10.1073/pnas.2024202118 (2021).
- Rausch, J. W., Capoferri, A. A., Katusiime, M. G., Patro, S. C. & Kearney, M. F. Low genetic diversity may be an Achilles heel of SARS-CoV-2. Proc Natl Acad Sci U S A 117, 24614-24616, doi:10.1073/pnas.2017726117 (2020).
- Chen, R. E. et al. Resistance of SARS-CoV-2 variants to neutralization by monoclonal and serum-derived polyclonal antibodies. Nat Med 27, 717-726, doi:10.1038/s41591-021-01294-w (2021).
- Starr, T. N. et al. Deep Mutational Scanning of SARS-CoV-2 Receptor Binding Domain Reveals Constraints on Folding and ACE2 Binding. Cell 182, 1295-1310 e1220, doi:10.1016/j.cell.2020.08.012 (2020).
- Wall, E. C. et al. Neutralising antibody activity against SARS-CoV-2 VOCs B.1.617.2 and B.1.351 by BNT162b2 vaccination. Lancet 397, 2331-2333, doi:10.1016/S0140-6736(21)01290-3 (2021).
- Motozono, C. et al. SARS-CoV-2 spike L452R variant evades cellular immunity and increases infectivity. Cell Host Microbe 29, 1124-1136 e1111, doi:10.1016/j.chom.2021.06.006 (2021).
- Grifoni, A. et al. SARS-CoV-2 human T cell epitopes: Adaptive immune response against COVID-19. Cell Host Microbe 29, 1076-1092, doi:10.1016/j.chom.2021.05.010 (2021).
- Tarke, A. et al. Comprehensive analysis of T cell immunodominance and immunoprevalence of SARS-CoV-2 epitopes in COVID-19 cases. Cell Rep Med 2, 100204, doi:10.1016/j.xcrm.2021.100204 (2021).
- Furutsuki, T. et al. Frequent transmission of cytotoxic-T-lymphocyte escape mutants of human immunodeficiency virus type 1 in the highly HLA-A24-positive Japanese population. J Virol 78, 8437-8445, doi:10.1128/JVI.78.16.8437-8445.2004 (2004).